Einleitung
In den letzten Jahren hat sich das Wissen über die menschliche Mikrobiota und ihre Auswirkungen auf Gesundheit und Krankheit erheblich verbessert. Viele medikamentöse Verfahren zielen darauf ab, das Mikrobiom eines Patienten zu manipulieren, indem neue Mikrobenstämme hinzugefügt, schädliche Stämme entfernt oder die Zusammensetzung des bestehenden Mikrobioms neu ausbalanciert wird (McChalicher & Aunins, 2022). Laktobazillen und Bifidobakterien sind Bakterien des menschlichen Magen-Darm-Trakts (GI), die traditionell als probiotische Nahrungsergänzungsmittel verwendet werden und auch als pharmazeutische Produkte klinisch geprüft werden. Für beide Bakteriengattungen wurden technologisch robuste Stämme isoliert und in industriellem Maßstab gezüchtet, nachdem sie lange Zeit in der Lebensmittelindustrie verwendet wurden. Studien zur menschlichen Darmmikrobiota haben jedoch andere Arten von Kommensalbakterien wie Akkermansia und Faecalibacterium hervorgehoben, die bei verschiedenen Krankheiten durchweg unterrepräsentiert sind (Andrade et al., 2020) und auf der Grundlage von Zell- und Tiermodellen möglicherweise ein diätetisches oder pharmazeutisches Potenzial haben (O'Toole et al., 2017). Während viele Aspekte eines lebenden mikrobiellen Arzneimittels, wie Wirksamkeit, Sicherheit und physiologische, genomische und metabolomische Eigenschaften, beträchtliche Forschungsarbeit erfordern, um das therapeutische Potenzial zu ermitteln, müssen auch andere oft übersehene, aber wesentliche Aspekte wie Herstellung, Lagerstabilität und Verabreichung untersucht werden, bevor eine breite praktische Anwendung möglich ist (Jimenez et al., 2019).
Viele kommensale Mikroorganismen im Magen-Darm-Trakt sind strikte Anaerobier mit anspruchsvollen Ernährungsbedürfnissen und stellen daher erhebliche neue Herausforderungen für die pharmazeutische Entwicklung dar, insbesondere bei der Herstellung, Lagerung und Verabreichung von Medikamenten (Andrade et al., 2020). Bei vielen interessanten GI-Arten sind die Ernährungsanforderungen der Organismen zunächst nur unzureichend bekannt, so dass für jeden neuen Organismus umfangreiche Forschungen im Bereich der Bioverfahrenstechnik durchgeführt werden müssen, um die Kultivierung der Produktionsziele zu ermöglichen. Eine zentrale Herausforderung bei der Entwicklung und Herstellung ist die Notwendigkeit, eine sauerstofffreie Umgebung aufrechtzuerhalten, die für die Lebensfähigkeit von obligaten Anaerobiern erforderlich ist (Mori et al., 2014). Obwohl automatisierte Systeme für die Bioprozessentwicklung sehr wertvoll sind, steigt die Komplexität der anaeroben Prozesskontrolle mit den zusätzlichen Verbindungen, die mit der Automatisierung verbunden sind.
Automatisierte anaerobe Probenahme
Numera® ist ein modulares Probenahmesystem, das flexibel konfiguriert werden kann, um eine kleinvolumige Hochfrequenz-Probenahme aus bis zu 16 Bioreaktoren oder nachgeschalteten Schwalltanks, eine zuverlässige Probenverarbeitung mit einer präzisen Verdünnung (Fehler ≤ 2 % SD) und einer einzigartigen Bandfiltrationstechnologie, eine gekühlte Probenlagerung im Fraktionssammlermodul und den Transfer zu mehreren Zell- und biochemischen Analysegeräten oder HPLC-Systemen zu ermöglichen (Abbildung 1). Darüber hinaus wird Numera® mit der leistungsstarken Lucullus®-Software für das Prozessinformationsmanagement geliefert, die eine digitale Integration der Geräte und Analysatoren, die Erfassung und zentrale Speicherung aller Prozessdaten sowie eine prozessweite Überwachung und Steuerung ermöglicht.

direkt zu den angeschlossenen Analysegeräten von Drittanbietern.
Die Numera®- und Lucullus®-Technologien werden standardmäßig in der vorgelagerten Bioprozessforschung eingesetzt, um Arbeitsabläufe zu automatisieren und zu digitalisieren und Forscher bei der automatischen Messung kritischer Prozessparameter (CPPs) und kritischer Qualitätsattribute (CQA) zu unterstützen, um Nährstoff- und andere Organismusanforderungen zu ermitteln. Hier werden Änderungen an der Standardinstallation von Numera® beschrieben, um die Kompatibilität mit strengen Anaerobiern zu ermöglichen. Um die Proben sanaerob zu verwalten, zu handhaben und zu halten, wurde das Numera®-System mit sterilem N2 anstelle von steriler Luft betrieben. Außerdem wurde den Fläschchen des Fraktionssammlermoduls eine sauerstoffabfangende Substratlösung zugesetzt, um die Probe vor der Anwesenheit von Umgebungssauerstoff zu schützen.
Material und Methoden
Aufbau der Kultivierung: Die Experimente wurden mit einem Wildtyp-Stamm der Blautia-Spezies aus der firmeneigenen Stammbibliothek von Seres Therapeutics durchgeführt, der mit dem Menschen verwandt ist. Die Kultivierung erfolgte in einem 1,0-L-DASGIP® Parallel Bioreactor System (Eppendorf) für den Einmalgebrauch. Der Bioreaktor wurde mit 1000 ml steril gefiltertem Medium (0,22 μm Filter) gefüllt, das für das Wachstum dieses Stammes optimiert wurde. Der gefüllte Bioreaktor wurde vor der Inokulation mit Stickstoffgas gespült, um gelösten Sauerstoff aus dem Bioreaktorsystem zu entfernen. Die Bioreaktorbedingungen umfassten einen Temperatursollwert von 33 °C, eine Rührgeschwindigkeit von 100 U/min und eine Sparringrate von 0,003 VVM Stickstoff. Der Bioreaktor verfügte über zwei getrennte Probenleitungen: eine für die manuelle Probenahme und die andere für das automatische Probenahmesystem Numera®. Ein Online-Gasanalysator wurde an den Abgasfilter des Bioreaktors angeschlossen, um den Sauerstoff- und Kohlendioxidausstoß während des Kultivierungsprozesses zu messen.
Aufbau der automatisierten Online-Probenahme: Das installierte Numera®-System verwendet eine pneumatische Bewegung der Proben unter Verwendung von Stickstoffgas (2,0 bar) anstelle einer typischen Installation, die Druckluft verwendet. Das Numera®-System wurde mit der Software Lucullus® (Securecell AG) gesteuert und mit den vorgegebenen Probenahmezeitpunkten auf der Grundlage der verstrichenen Kultivierungszeit konfiguriert. Zu jedem Zeitpunkt wurden die vom Numera®-Multiplexermodul entnommenen Proben in zwei Probenfraktionen aufgeteilt: 1,0 mL Vollbrühe wurde direkt in das Fraktionssammelmodul in Glasfläschchen mit 30 μL Sauerstofffängersubstrat (Oxyrase, Oxyrase®Inc.) abgegeben, und 1,0 mL geklärte Brühe wurde zunächst mit dem Filtrationsmodul aufbereitet und dann in Glasfläschchen im Fraktionssammler abgegeben. Beide Probenfläschchentypen werden auf einem gekühlten Gestell bei 4°C aufbewahrt.
Manuelle Offline-Probenahme: Offline-Proben wurden aus dem Bioreaktor entnommen. An jedem Probenahmepunkt wurden 5 ml Kulturbrühe manuell entnommen und sofort zur Verarbeitung in eine anaerobe Kammer überführt. Die Proben wurden auf koloniebildende Einheiten (CFUs, Gehirn/Herz-Infusionsagar), den Offline-pH-Wert und die optische Dichte (600 nm) untersucht.
Probenanalyse von automatisierten Proben: Ganze Brüheproben, die auf dem Fraktionssammelmodul des Autosamplersystems Numera® aufbewahrt wurden, wurden in eine anaerobe Kammer überführt und wie oben beschrieben auf KBEs analysiert. Die filtrierten Proben wurden mit einem Ionenchromatographiesystem (Thermo Scientific™, Dionex™ ICS-6000) auf die prozessinterne Zuckerverwertung analysiert.
Ergebnisse und Diskussion
Seres Therapeutics hat eine Stammbibliothek für menschliche Gastroenterologen zusammengestellt, die viele obligate Anaerobier mit unterschiedlichen Ernährungsanforderungen und Umweltempfindlichkeiten umfasst. Die Sauerstoffexposition ist für diese Isolate oft toxisch, wie es für gut untersuchte obligate Anaerobier wie acetogene Bakterien, Methanogene und Clostridien typisch ist (Rigottier-Gois, 2013). Um das in diesem Bericht beschriebene Numera-System zu testen, wurde ein eigener Blautia-Stamm auf der Grundlage der zuvor ermittelten Empfindlichkeit gegenüber Sauerstoffexposition und der internen phänotypischen Bewertung ausgewählt. Um die Sauerstoffempfindlichkeit des in diesen Studien verwendeten Blautia-Stammes zu ermitteln, wurden anaerob gewachsene Kulturproben zwei Bedingungen ausgesetzt: einer hypoxischen Umgebung (0,4 % Sauerstoff) für 180 Minuten oder Luft (~21 % Sauerstoff) für 20 Minuten. Die Exposition an der Luft wirkte sich nachteilig auf die Lebensfähigkeit aus und führte zu einer 10.000-fachen Verringerung des Titers der lebensfähigen Zellen des Blautia-Stammes im Vergleich zur Exposition in der hypoxischen Umgebung (Abbildung 2). Dieser Phänotyp ist typisch für die sauerstoffempfindlichen Stämme in der Seres-Bibliothek und stellt sicher, dass dieser Stamm ein geeigneter Modellorganismus für Studien ist, in denen die Fähigkeit des angepassten Systems zum Schutz vor Sauerstoffexposition überprüft wird. Die Balken in Abbildung 2 zeigen die Bandbreite der in der Seres-Bibliothek beobachteten Phänotypen in Bezug auf hypoxische und vollständig aerobe Expositionen. Diese Daten zeigen, dass es wichtig ist, die Exposition von obligaten Anaerobiern gegenüber Sauerstoff während der Kultivierung zu minimieren und repräsentative Proben mit begrenzter Sauerstoffexposition zu erhalten, um zuverlässige Messungen der Zelllebensfähigkeit für Studien zur Bioprozessentwicklung zu gewährleisten.

Da die Exposition gegenüber Sauerstoff eine tiefgreifende Auswirkung auf die Lebensfähigkeit haben kann, haben wir untersucht, ob die Aufbewahrung der Proben in Fläschchen, die zuvor mit einer sauerstoffbindenden Substratlösung gefüllt wurden, die Lebensfähigkeit während der Aufbewahrungsschritte schützen würde. Tabelle 1 zeigt den Versuchsplan, mit dem die Auswirkungen der Aufbewahrungsbedingungen (anaerob vs. aerob und RT vs. 4°C) und der Aufbewahrungsdauer auf die Lebensfähigkeit der vegetativen Zellen (Titer) der Proben getestet wurden. Es wurden vier Stämme getestet: zwei mit hoher Sauerstoffempfindlichkeit (darunter der oben beschriebene Blautia-Stamm) und zwei mit geringer Sauerstoffempfindlichkeit. Die Ergebnisse werden als Log-Reduktionsfaktor (LRF) in logarithmischer Skala zwischen dem Titer ohne Haltezeit und dem Titer mit Haltezeit verglichen.
Um eine zuverlässige Entscheidungsfindung in der zukünftigen Bioprozessforschung und -entwicklung zu ermöglichen, wurde ein oberer log-Reduktionsfaktor von 0,5 log10 als Auslegungskriterium für die Lagerbedingungen gewählt. Um die Konstruktion und den Betrieb der Anlage zu erleichtern, sollten die Proben in einer vollständig aeroben Umgebung gelagert werden. Auf der Grundlage der Ergebnisse der konzipierten Studie (Daten nicht gezeigt) ist die Lagerung in einer aeroben Umgebung für mindestens 3 Stunden selbst für die empfindlichsten Stämme möglich, wenn die Proben bei 4 °C und in Gegenwart des Sauerstoff abfangenden Substrats aufbewahrt werden. Bei Stämmen, die sehr empfindlich auf Sauerstoff reagieren, erhöhen längere Lagerungszeiten die log-Reduktionsfaktoren über 0,5 log10 hinaus, und bei Proben, die zu einem früheren Zeitpunkt der Wachstumsphase entnommen wurden, wurde ein höherer LRF beobachtet als bei Proben, die in einer späteren Wachstumsphase des Kultivierungsprozesses entnommen wurden. Weitere Untersuchungen sind erforderlich, um eine allgemeine Schlussfolgerung für ein breiteres Spektrum von obligaten Anaerobiern ziehen zu können. Bei den späteren Experimenten, über die in dieser Studie berichtet wird, enthielten die Aufbewahrungsgefäße des Numera®-Fraktionssammlers 30 μl Oxyrase-Substratlösung pro 1 mL Probenmenge, basierend auf den experimentellen Ergebnissen.
Die Leistung des Numera®-Systems zur Überwachung des sauerstoffempfindlichen Blautia-Stamms während der Kultivierung in Bioreaktoren wurde sowohl während der exponentiellen als auch der stationären Wachstumsphase bewertet. Manuelle und automatische Numera®-Proben wurden nach 12, 14, 16, 18 und 20 Stunden anaerober Kultivierung entnommen (siehe Abschnitt Material und Methoden), um einen direkten Vergleich der beiden Methoden zu ermöglichen. Die manuellen Proben wurden direkt nach der Probenahme auf den Titer (KBE/ml) hin analysiert (Angabe in logarithmischer Skala), während die automatisierten Numera®-Proben auf dem Fraktionssammler bei 4 °C in Oxyrase-haltigen Fläschchen aufbewahrt und nach einer Haltezeit von 2 oder 4 Stunden nach der Probenahme analysiert wurden (Abbildung 3).

Die roten Balken stehen für manuell entnommene Proben, die sofort nach der Entnahme analysiert wurden (Sample Hold Time = 0), während die blauen Balken für automatisierte Numera®-Proben stehen, die nach 2 bzw. 4 Stunden Sample Hold Time analysiert wurden. Jeder Fehlerbalken ist mit 1 Standardfehler vom Mittelwert konstruiert.
Die automatisierten Numera®-Proben zeigten weniger als die Hälfte des Log10-Abfalls nach einer 2- oder 4-stündigen Haltezeit während der späten exponentiellen bis zur stationären Phase (Abbildung 4). Zu allen Zeitpunkten außer dem anfänglichen Zeitpunkt der mittleren exponentiellen Wachstumsphase bei 12,5 Stunden lag der Unterschied zwischen den Probenahmeverfahren innerhalb des 0,5 log10 log-Reduktionsfaktors, der in der oben genannten Studie zur Entwicklung der Lagerbedingungen angestrebt wurde. Am 12,5-Stunden-Zeitpunkt war der Unterschied mit einem beobachteten log-Reduktionsfaktor von 0,8 log10 größer, allerdings nach einer Haltedauer von 4 Stunden statt der vorgesehenen Lagerungsgrenze von 3 Stunden.
Zusätzlich zur Lebensfähigkeitsanalyse wurden mit dem Numera®-System gefilterte Proben nach einer Kultivierungsdauer von 6, 8, 9, 10, 11 und 12 Stunden gesammelt. Diese Proben wurden anschließend verwendet, um die Schätzung der Zuckerverbrauchsrate während des exponentiellen Wachstums zu analysieren (Abbildung 5). Diese gefilterten Proben wurden während der gesamten Kultivierungsdauer vor der Analyse bei 4°C auf dem Fraktionssammler aufbewahrt.

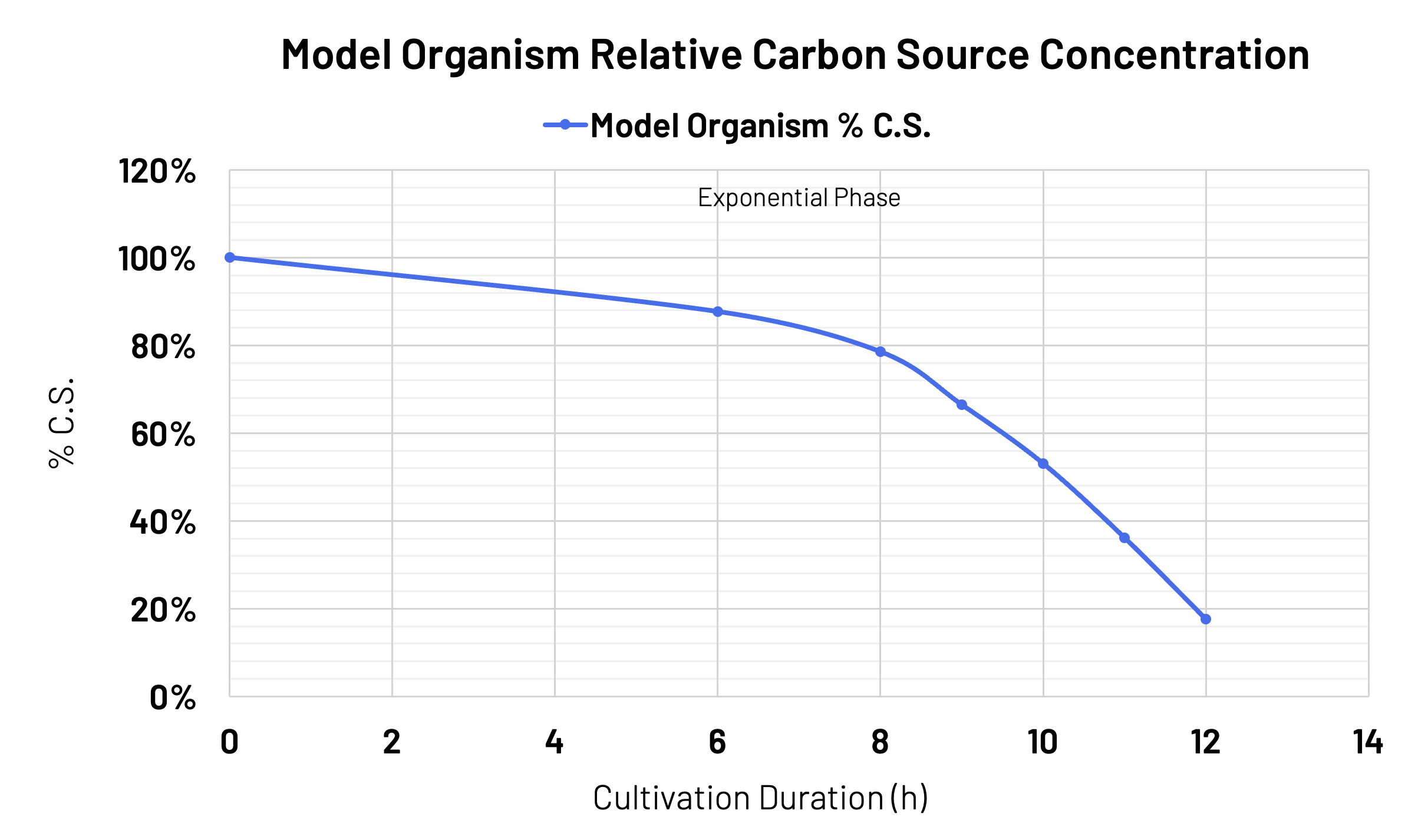
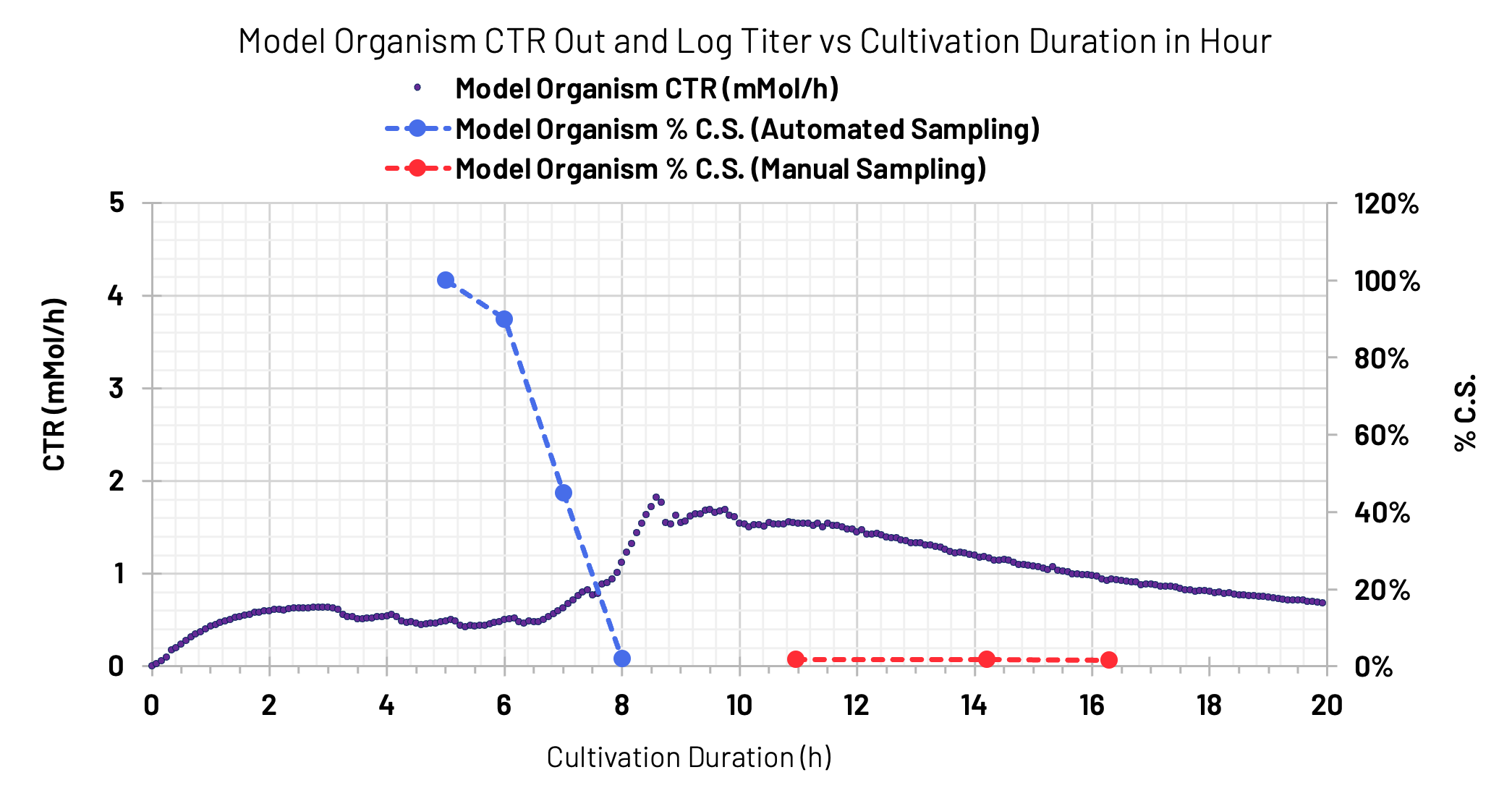
Die Eignung des Numera®-Systems, einschließlich der entwickelten Probenahmemethode, wurde für ganztägige Probenahmen, einschließlich unbesetzter Nachtperioden, mit dem Modellstamm Blautia bewertet. Die automatisierten Numera®-Proben wurden auf der Grundlage der verstrichenen Fermentationszeit 3, 4, 5, 6, 7 und 8 Stunden nach der Inokulation des Bioreaktors geplant. Zusätzlich wurden manuelle Proben 11, 14 und 16 Stunden nach der Inokulation genommen. Die Haltezeit der automatisierten Numera®-Proben variierte je nach Probenahmezeitpunkt, mit einer maximalen Haltezeit von sechs Stunden für die erste dreistündige Probe und kürzeren Haltezeiten für jede weitere Probe. Alle automatisch entnommenen Proben wurden neun Stunden nach Beginn der Kultur auf Lebensfähigkeit und Restkohlenstoffquelle analysiert. Zusätzlich wurden Abgasdaten gesammelt und die Kohlendioxid-Transferrate (CTR) aus dem Kohlendioxidgehalt im Abgas und der Durchspülungsrate berechnet. In Abbildung 6 sind die Ergebnisse der Lebensfähigkeitsmessungen dargestellt. Sie zeigen eine exponentielle Wachstumsrate von 0,87 h-1 während der Probenahme über Nacht bis zum 8-Stunden-Probenahmezeitpunkt. Die Lebensfähigkeitsmessung der Proben nach 10 Stunden nach der Inokulation zeigt den Beginn der stationären Phase. Diese Beobachtungen werden durch die CTR- und Restkohlenstoffquellenmessungen in Abbildung 7 bestätigt. Die CTR-Daten zeigen ein exponentielles Wachstum von etwa 5 bis 9 Stunden nach der Inokulation und dann den Beginn der stationären Phase, was dem vollständigen Verbrauch der Kohlenstoffquelle entspricht.
Das automatisierte Numera®-System ermöglichte die zuverlässige Sammlung eines umfassenden Satzes von
Proben auch außerhalb der normalen Arbeitszeiten. Es hat sich gezeigt, dass die Analyse dieser Proben, einschließlich der Analyse der Zelllebensfähigkeit, die Online-Kultivierungsdaten ergänzt. Diese Datensätze sind ausreichend, um die weitere Entwicklung optimierter Mediumrezepte für eine kontinuierliche Prozessverbesserung, die Implementierung von Fed-Batch-Strategien zur weiteren Produktionssteigerung oder den Nachweis der Robustheit von Prozesskontrollen über eine Reihe von Wiederholungsversuchen zu ermöglichen.

Jeder Fehlerbalken ist unter Verwendung von 1 Standardfehler vom Mittelwert konstruiert.
Schlussfolgerung
Die hier vorgestellten Arbeiten haben bestätigt, dass das Numera®-System ein geeignetes Instrument für die automatisierte Probenahme bei der Kultivierung von obligaten Anaerobiern sein kann, einschließlich der genauen Bestimmung von lebensfähigen Titern für Proben nach einer Haltezeit von bis zu 3 Stunden in aerober Umgebung. Während der anfänglichen Entwicklung der Probenahmemethode wurden vergleichbare Probenmessungen wie bei der manuellen Probenahme durch die kombinierte Verwendung von Stickstoffgas anstelle von Luft für die Systempneumatik, die Verwendung eines sauerstoffabfangenden Substrats in den Sammelgefäßen zur Probenkonservierung und die Kühlung der Sammelgefäße auf 4 °C erzielt. Während im Allgemeinen Haltezeiten von bis zu 6 Stunden für Proben, die während des Kultivierungsprozesses entnommen wurden, für die hier getesteten obligaten Anaerobier als geeignet angesehen wurden, können die optimalen Bedingungen von Stamm zu Stamm variieren und müssen möglicherweise empirisch ermittelt werden. Größere Unterschiede bei den Lebensfähigkeitsmessungen wurden bei Kulturen in der exponentiellen Phase beobachtet, wobei sich der Unterschied mit dem Fortschreiten der Kulturen in die späte exponentielle und stationäre Phase verringert. Systematische Unterschiede zwischen manuellen und automatisierten Proben können in Abhängigkeit von inhärenten Stammcharakteristika einschließlich des Zellwachstumszyklus beobachtet werden. Weitere Untersuchungen sind erforderlich, um festzustellen, ob diese Unterschiede auf eine unterschiedliche Sauerstoffempfindlichkeit, Stoffwechselaktivität oder eine andere physiologische Eigenschaft dieser Zellen in der jeweiligen Wachstumsphase zurückzuführen sind. Die Qualität der in dieser Studie gewonnenen Daten eignet sich für viele Forschungs- und Entwicklungsaktivitäten im Bereich der Bioprozesse, einschließlich Studien zur frühen Medienentwicklung, zur Optimierung der Fed-Batch-Prozesssteuerung oder zur Identifizierung von wachstumslimitierenden Nährstoffen. Weitere Verbesserungen der Systemkonfiguration, wie z. B. die Platzierung des Fraktionssammelmoduls in einem sauerstofffreien Isolator, können die Probenkonservierung für sauerstoffempfindliche Mikroben weiter verbessern. Die Konfiguration des Numera®-Systems mit Online-Messungen der Lebensfähigkeit könnte zusätzlich die Auswirkungen der Probenhaltezeiten verringern.
Auch wenn es noch Möglichkeiten für weitere Forschung und Verbesserungen gibt, ermöglicht der hier beschriebene Aufbau eine automatisierte Probenahme von obligaten anaeroben Kulturen und liefert
vergleichbare Ergebnisse für die Messung der Lebensfähigkeit und die Erhaltung von Proben, die für die Messung der Substratkonzentration und der optischen Dichte geeignet sind. Wichtig ist, dass die Haltezeiten ausreichend sind, um längere Zeiten ohne Beteiligung des Bedieners zu ermöglichen. In Verbindung mit anderen Online-Messungen, wie der Kohlendioxidproduktion, ermöglichen die aus diesen Proben gewonnenen Daten eine konsistente Interpretation des Kultivierungsprofils und damit eine weitere Optimierung oder Robustheitsanalyse.
Wichtige Ergebnisse
-
Sauerstoffexposition ist oft schädlich für Bakterien, die mit dem Menschen in Kontakt kommen, einschließlich Firmicutes, und führt unter den getesteten Bedingungen zu einer 10.000-fachen Reduzierung der Zellkonzentration nach 20 Minuten.
-
Die kombinierten Wirkungen von N2-Gas für die Pneumatik, die Vorladung von Sauerstofffängersubstrat
Die kombinierte Wirkung von N2-Gas für die Pneumatik, der Zugabe von Sauerstofffängersubstratlösung in die Fraktionssammelgefäße und dem Halten der Gefäße bei 4°C im Fraktionssammelmodul ermöglichte das stabile Halten lebensfähiger obligater Anaerobierproben. -
Die Stabilität der Proben wurde anhand von Messungen des Titers lebensfähiger Zellen bis zu 6 Stunden lang aufrechterhalten
-
Numera® ermöglichte eine zuverlässige Probenahme in Zeiten, in denen kein Bediener anwesend war. Die Kombination von stabilen Proben für die Messung der Anzahl lebensfähiger Zellen und die Verwendung von geklärten Brüheproben für die Messung von Restsubstraten. Zusätzlich zur Online-Messung der Kohlendioxidentwicklung ermöglicht Numera® eine robuste Untersuchung der Kulturleistung und liefert ausreichend Daten für eine fortlaufende Optimierung und Robustheitsbewertung.
Referenzen
- Andrade, J. C., Almeida, D., Domingos, M., Seabra, C. L., Machado, D., Freitas, A. C., & Gomes, A. M. (2020). Kommensale obligate anaerobe Bakterien und Gesundheit: Production, Storage, and Delivery Strategies. Frontiers in Bioengineering and Biotechnology, 8. https://www.frontiersin.org/journals/bioengineering-and-biotechnology/articles/10.3389/fbioe.2020.00550
- Jimenez, M., Langer, R., & Traverso, G. (2019). Microbial therapeutics: New opportunities for drug delivery. Journal of Experimental Medicine, 216(5), 1005-1009. https://doi.org/10.1084/jem.20190609
- McChalicher, C., & Aunins, J. (2022). Drugging the microbiome and bacterial live biotherapeutic consortium production. Current Opinion in Biotechnology, 78, 102801. https://doi.org/10.1016/j.copbio.2022.102801 O
- 'Toole, P. W., Marchesi, J. R., & Hill, C. (2017). Probiotika der nächsten Generation: Das Spektrum von Probiotika zu lebenden Biotherapeutika. Nature Microbiology, 2(5), 17057. https://doi.org/10.1038/nmicrobiol.2017.57
- Rigottier-Gois, L. (2013). Dysbiose bei entzündlichen Darmerkrankungen: die Sauerstoffhypothese. The ISME Journal, 7(7), 1256-1261. https://doi.org/10.1038/ismej.2013.80











